Aprendizaje Supervisado

El aprendizaje supervisado es una técnica usada en minería de datos, en la que se genera una función de pronóstico a partir del entrenamiento sobre datos etiquetados, es decir, aprendemos a partir de casos reales y extrapolamos el resultado a los casos futuros.
Se dice que es supervisado porque, previamente debe existir una clasificación o etiquetado de los datos que es lo que aporta el conocimiento. Otros modelos como las redes neuronales no necesitan este conocimiento previo para realizar el pronóstico.
Comenzar un modelo
Lo primero es dividir la muestra en dos conjuntos, uno de entrenamiento y otro de prueba o test. Con los datos de entrenamiento ordenados convenientemente obtenemos un conjunto de vectores o pares de entrada-salida.
Las entradas son las variables conocidas que usaremos para hacer el pronóstico. La salida es la variable dependiente que queremos pronosticar o clasificar.
Los algoritmos “aprenden” de los datos de entrenamiento y ese conocimiento obtenido del estudio de los datos previos clasificados lo representaremos en forma de modelo matemático. Cuando metemos una nueva entrada en el modelo, nos pronostica la salida más probable.
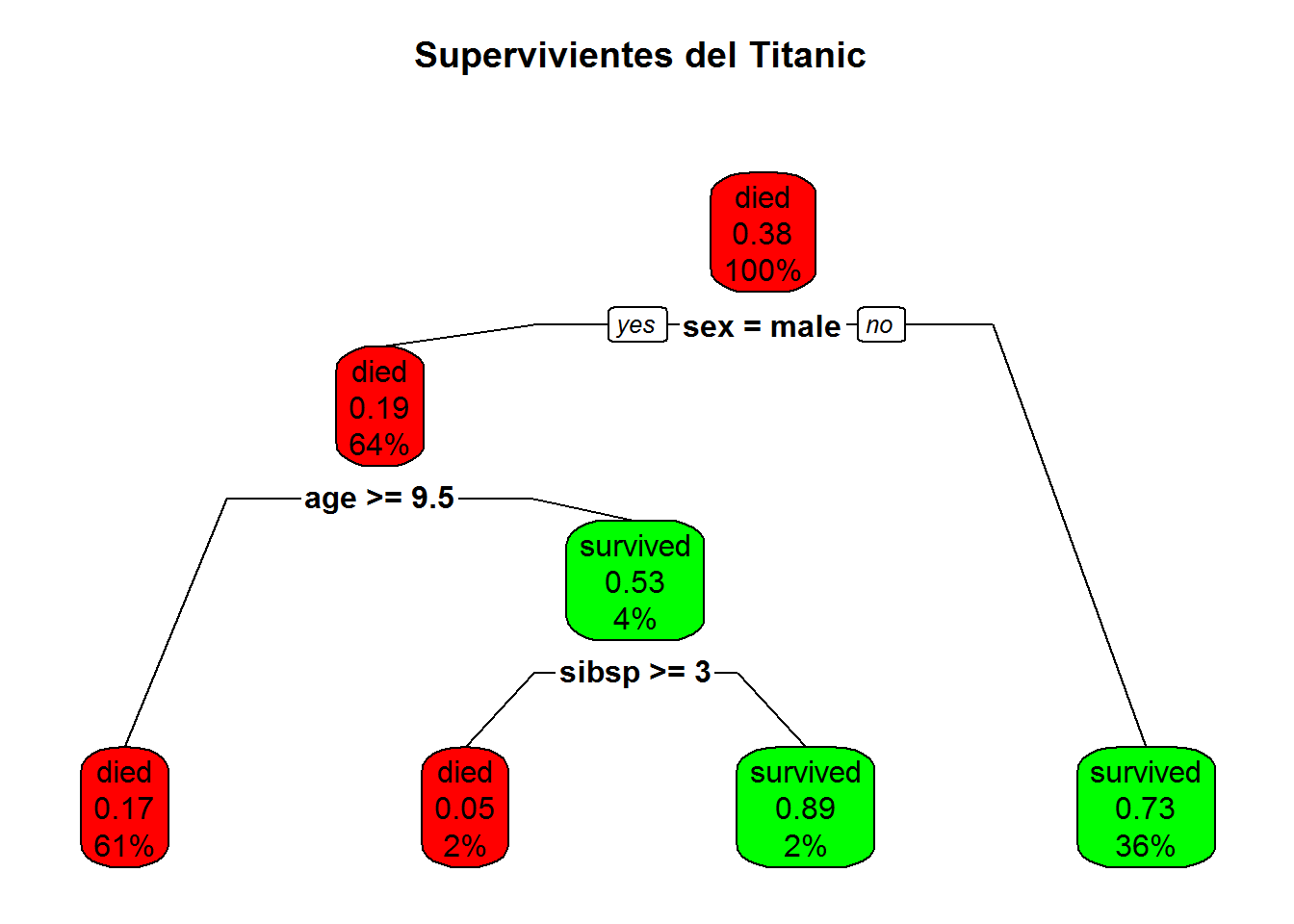
Ejemplo de arbol de decisión
Los modelos de aprendizaje supervisado, se denominan habitualmente modelos de clasificación ya que tratan de agrupar los valores en conjuntos con características semejantes, y la respuesta es el grupo al que creen que pertenece el hecho definido en la entrada.
Principales algoritmos de aprendizaje supervisado
Existen diferentes algoritmos que abordan el problema de aprendizaje supervisado y técnicas de minería de datos en R, pero los más habituales son los 5 siguientes:
- knn ((k-Nearest Neighbour Classification).
- naive bayes o algoritmo ingenuo
- regresión logística binaria
- árboles de decisión
- bosques de clasificación
He realizado un manual básico de aprendizaje supervisado en R en el que describo estos 5 algoritmos y alguno más de forma práctica y que puedes ver libremente aquí.
Solo como recordatorio, pongo también el enlace a mi libro Manual de R para el científico de datos, cuya descarga es completamente gratis.
Tabla resumen de uso
Aunque la información detallada está en el manual, aportamos en la siguiente tabla un resumen de uso práctico de cada modelo que puede venir bien tener a mano, indicando el paquete o librería de R y la función de pronóstico principal para cada uno:
- knn
- library(class)
m<-knn(train = tabla_train, test = tabla_test, cl = tabla_train$col_clasificadora)- el resultado de la función es el tipo de clase
clal que pertenecen los datos de la tabla_test - Para optimizar los resultados el modelo necesita estandarizar las variables normalizar distancias
- scale(tabla)
- naivebayes
- library(naivebayes)
m<-naive_bayes(var_dependiente ~ var1 + var2..., data = tabla_datos, laplace = n)laplace es opcional y agrega n casos para cada supuesto combinatorio.predict(m)predicción sobre toda la tabla origenpredict(m,h,type="prob")siendohun hecho endata.frame.type = "prob"muestra probabilidad de cada clase de salida, si se omite type=, el resultado es la clasificación más probable.
- naivebayes_e1071
- library(e1071)
m<-naiveBayes(var_dependiente ~ ., data = tabla_datos_train)predict(m)predicción sobre toda la tabla origentabla_datos_train, ,type="prob"opcionalpredict(m,tabla_datos_test)
- Regresión logistica
glm- library(stats), cargada por defecto con R base.
m_glm<-glm(y ~ x1 + x2 + x3,data = misdatos_train, family = "binomial")m_glm<-glm(y ~ x1*x2, data = misdatos_train, family = "binomial")para variables con impacto combinado, efecto conjunto exponencial sobre la variable dependiente.- probabilidad:
prob <- predict(m_glm, misdatos_test, type = "response")usar tipo response - umbral de predicción:
pred <- ifelse(prob > 0.50, 1, 0) summary(m_glm)
- Optimizacion de regresión logistica
glm- Se usa para hallar el mejor modelo de pronóstico, definiendo un inicio y fin de modelos:
1.Modelo sin predictores:null_model <- glm(var1 ~ 1, data = misdatos, family = "binomial")2.Modelo completo:full_model <- glm(var1 ~ ., data = misdatos, family = "binomial")- función step():
step_model <- step(null_model, scope = list(lower = null_model, upper = full_model), direction = "forward")
- función step():
summary(step_model)da el resultado de las pruebas con el mejor modelo- Para estimar la probabilidad:
step_prob <- predict(step_model, type = "response")
- Se usa para hallar el mejor modelo de pronóstico, definiendo un inicio y fin de modelos:
- Árboles de decisión
- los datos origen a clasificar deben ser factores o estar categorizados. No es conveniente usar datos continuos.
library(rpart)- Hay que definir una poda
poda<-rpart.control(cp= 0.2, maxdepth = 30, minsplit = 20)que limita el árbol, lo más común es usar solo cp=0.2. m_tree<-rpart(var_dependiente ~ ., data = tabla_datos_train, control = poda)predict(m_tree, h,type = "class")predicción sobre el hechohpredict(m,tabla_datos_test)predicción sobre los datos de test- Se puede dibujar el árbol con rpart.plot:
rpart.plot(m_tree, type = 2, clip.right.labs = FALSE,branch = .6,box.palette = c( "red","green"),
main = "Ejemplo de arbol \n lalala")
- Bosques aleatorios.
- Los bosques son sumas de modelos de árboles. Por lo que es mejor no usar datos continuos sino categorizados.
library(randomForest)m_bosque <- randomForest(var_dependiente ~ ., data = tabla_datos_train[complete.cases(tabla_datos_train),], ntree = 100 )Hemos excluido todos los posibles NA, pero podría hacerse previamente unimputede estos datos.predict(m_bosque)predicción sobre toda la tabla origen
predict(m_bosque,tabla_datos_test), predicción sobre los datos de test- Pintamos la evolución del modelo y vemos a partir de qué numero de arboles los resultados son homogéneos:
plot(m_bosque)

